Schätzungen zufolge ist die Hälfte der Menschheit mit Helicobacter pylori infiziert. Je nach Region mehr oder weniger und je nach Antibiotika-Verfügbarkeit mehr oder weniger resistente Stämme. Häufig erfolgt die Infektion mit H. pylori bereits im frühen Kindesalter. Aufgrund der hohen Hygienestandards ist in Mitteleuropa die Inzidenz rückläufig jedoch steigen die Resistenzen.
Der H. pylori Nachweis erfolgt meistens mittels Bakterienkultur auf der Agarplatte, die auch eine Resistenztestung mittels Antibiogram ermöglicht. Oft wächst die Kultur nicht an und dann ist auch keine Resistenztestung möglich. Hier sollten molekulargenetische PCR Testsysteme zum Einsatz kommen, die Resistenzen nachweisen können, vor allem wenn der kulturelle Nachweis versagt. Üblicherweise dient als klinische Probe die Magenbiopsie nach einer Gastroskopie. Was die meisten nicht wissen: Moderne PCR-Systeme können auch H.pylori und dessen Claritromycin-Resistenz direkt aus Stuhl-Proben nicht invasiv nachweisen. Die Magenbiopsie ist alles andere als angenehm, wie ich selbst erleben “durfte”. Auch war das “Ergebnis” negativ; wohlwissend, dass bei weitem nicht jede Kultur anwächst.

Wenn aber die Symptome bleiben, dann kommt der Patient wieder und dann das gleiche unangenehme Prozedere mit der Gastroskopie von vorne. Rezidive sind keine Seltenheit und spätestens da ist es höchste Zeit für eine PCR Testung, die auch die Claritromycin (CLA)-Resistenzen erkennen.
Ein Fallbeispiel aus der Praxis hilft uns hier viel weiter: In einem Pathologie-Labor wird routinemäßig eine H. pylori PCR aus FFPE Schnitten durchgeführt. Parallel werden die Magenbiopsien eingeschickt und kultiviert.
Die Laborbefunde ergaben diskrepante Ergebnisse:
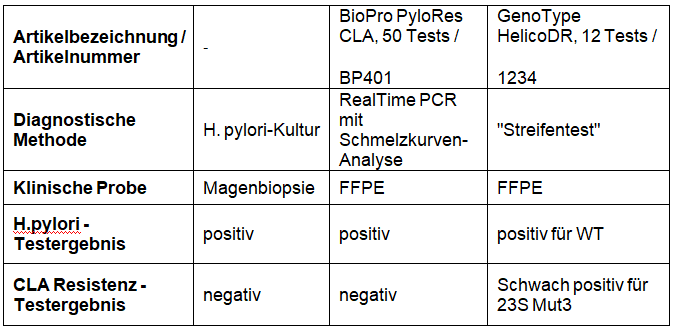
Eine Wiederholung ergab die gleichen Ergebnisse bei allen Testsystemen. Nachdem bei GenoType HelicoDR nur eine Bande für die CLA Resistenz ganz schwach positiv war, hätten wir ein Artefakt oder eine Laborkontamination vermutet; jedoch waren die Ergebnisse reproduzierbar!
Schließlich wurde die fragliche Probe bei BioProducts im Labor nachgetestet: und tatsächlich – eine schwach positive Resistenz bei CLA. Aufgereinigt wurde der FFPE Schnitt bei uns mit dem MagCore FFPE OneStep Kit. Bei uns konnte eine Kontamination ausgeschlossen werden und das Ergebnis war wieder reproduzierbar. Spannend: Weder in der Kultur konnte der im Hintergrund mickrig schwach wachsende resistente H.pylori Stamm erkannt werden noch mittels RealTime PCR war bei der Schmelzkurve ein entsprechender Peak erkennbar. In diesem Fall war die PCR mit anschließender reverser Hybridisierung der sogenannte “goldene Standard”.